이번에는 전처리 과정으로 넘어가보자
앞에서 다양한 형태의 EDA와 데이터프레임 통합 과정을 거친 덕분에 전처리는 상대적으로 수월하게 진행 가능해졌다.
다루게 될 df_norm 데이터 형태부터 살펴보자

여기에서 결과에 해당되는 Pres_DEM, Pres_REP, Gov_DEM, Gov_REP는 입력에서 제외하고, 값이 NaN인 데이터들도 drop처리를 해주도록 하자
df_norm.dropna(inplace = True)
X = df_norm.drop(['Pres_DEM', 'Pres_REP', 'Gov_DEM', 'Gov_REP'], axis = 1)
y = df_norm['Pres_DEM']
위 컬럼에서 보듯, 해당되는 모든 변수들이 수치형 데이터이기 때문에
표준화처리도 추가작업 없이 진행해주면 된다.
from sklearn.preprocessing import StandardScaler
# StandardScaler를 이용해 수치형 데이터를 표준화하기
scaler = StandardScaler()
scaler.fit(X)
X_scaled = scaler.transform(X)
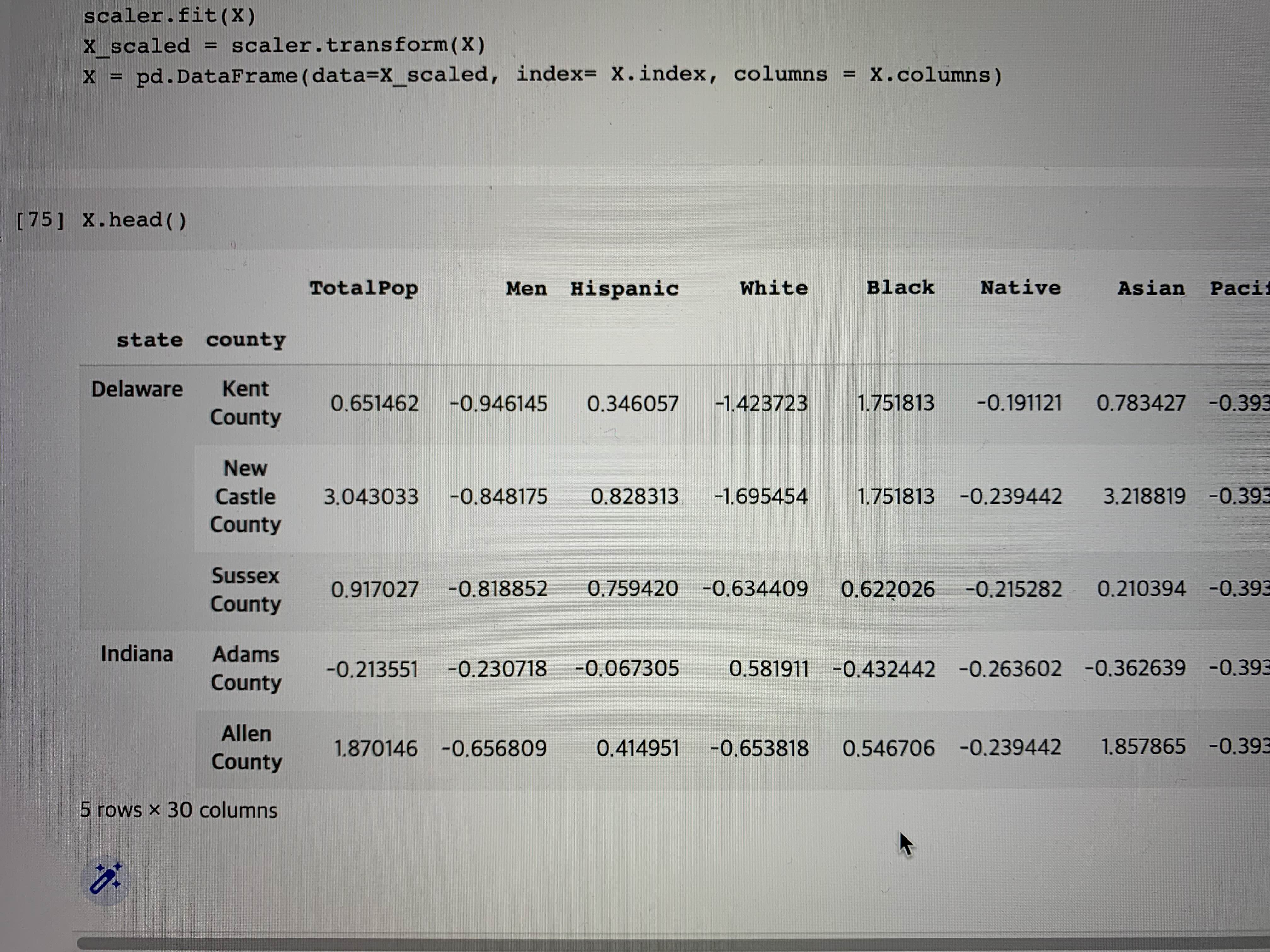
X = pd.DataFrame(data=X_scaled, index= X.index, columns = X.columns)
X가 모든 컬럼에 대해 표준화처리가 잘 된 것을 확인할 수 있다.
이후 train과 test 데이터 분리 역시 이전과 동일한 과정이다.
from sklearn.model_selection import train_test_split
# train_test_split() 함수로 학습 데이터와 테스트 데이터 분리하기
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size = 0.3, random_state = 1)
이전까지의 강의들에서는 이후 바로 모델링 과정으로 넘어갔었는데
이번 강의에서는 PCA를 통해 데이터 전처리를 추가로 진행하는 과정을 가진다.
우선 코드 및 결과와 함께 PCA에 대해 자세히 정리해보자
from sklearn.decomposition import PCA
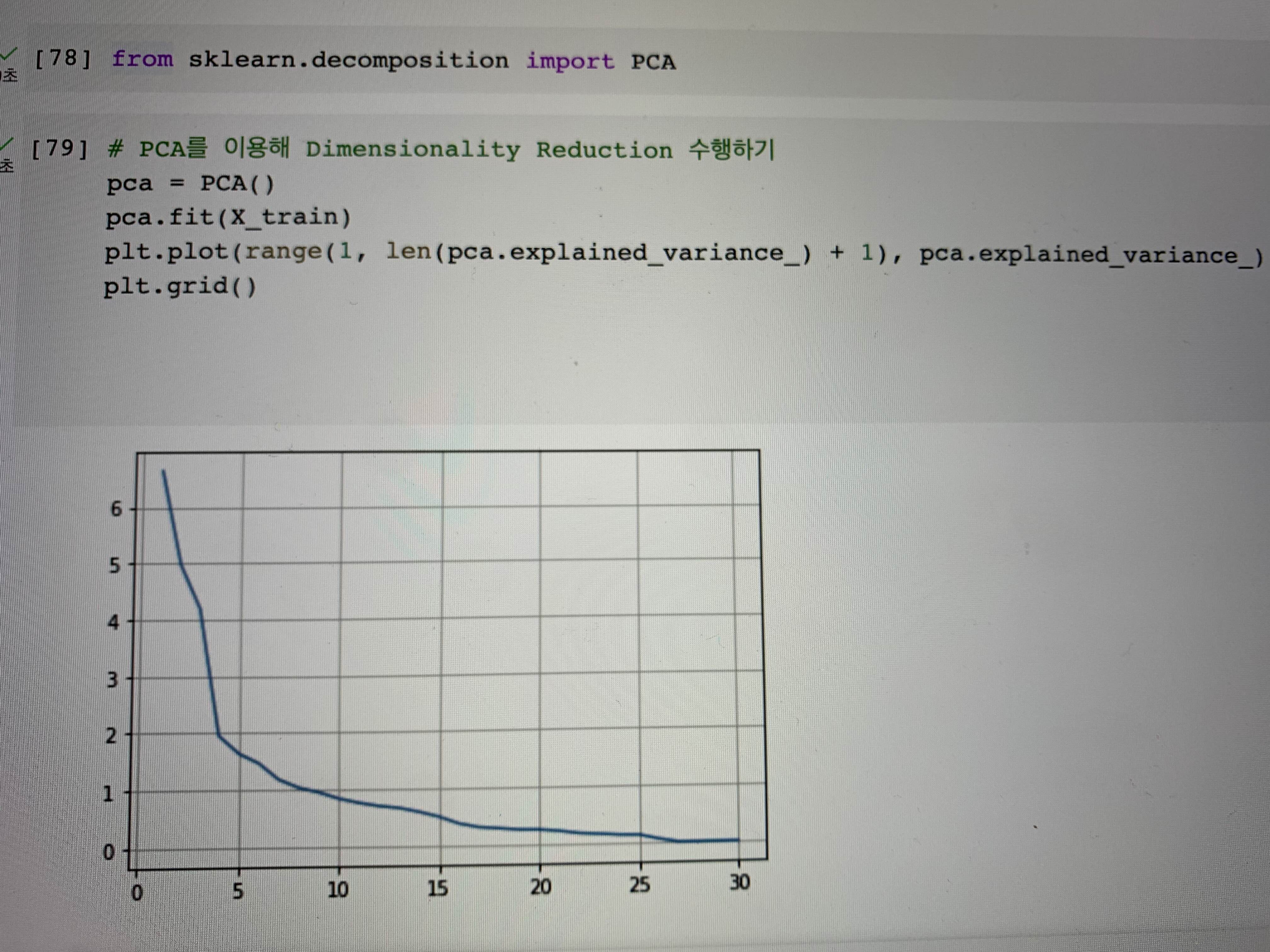
# PCA를 이용해 Dimensionality Reduction 수행하기
pca = PCA()
pca.fit(X_train)
plt.plot(range(1, len(pca.explained_variance_) + 1), pca.explained_variance_)
plt.grid()
PCA(Principal Component Analysis)는 고차원 데이터를 저차원으로 축소하는 기법 중 하나로
세부 파라미터 설정값들은 아래와 같다.
- n_components: 축소할 차원 수. 기본값은 None으로, 이 경우에는 모든 주성분을 유지한다.
- whiten: True로 설정하면 고유벡터가 서로 직교하게 만들고 분산을 1로 만들어 특징 벡터를 흰색으로 만들어준다. 기본값은 False
- svd_solver: SVD 알고리즘을 지정해준다. 'auto', 'full', 'arpack', 'randomized' 중 하나를 선택할 수 있으며, 기본값은 'auto'로 되어있다.
- random_state: 난수 발생 시드 값으로, 기본값은 None
또한, PCA를 적용한 결과는 fit_transform 메서드를 사용하여 계산이 가능한데 이 때, 분석 대상 데이터를 입력값으로 사용하면 PCA로 변환된 데이터가 반환된다.
# 코드 예시
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
reduced_data = pca.fit_transform(data)위 코드에서 n_components를 2로 지정하여 데이터를 2차원으로 축소하고, fit_transform 메서드를 사용하여 분석 대상 데이터 data를 변환한 결과를 reduced_data에 저장한다.
추가로 PCA를 통해 구한 주성분은 components_ 속성으로 확인 가능ㅏ다. explained_variance_ratio_ 속성을 통해 각 주성분이 전체 분산에서 차지하는 비율을 확인할 수 있다.
마지막으로 PCA 결과를 시각화하기 위해서는 matplotlib 라이브러리를 사용하여 산점도를 그릴 수 있다.
## 예시
# 주성분 비율로 확인하기
components = pca.components_
explained_variance_ratio = pca.explained_variance_ratio_
# 산점도로 표현하기
import matplotlib.pyplot as plt
plt.scatter(reduced_data[:, 0], reduced_data[:, 1])
plt.show()
PCA를 사용하여 데이터의 차원을 축소하면서 데이터의 구조를 최대한 유지하여 적은 수의 주성분으로 구성된 새로운 데이터를 구상할 수 있다. 이를 통해 고차원 데이터를 시각화하거나 분석에 원할히 활용할 수 있게 된다.
다시 실제 데이터로 돌아와서 결과를 살펴보자
위 그래프의 추이를 살펴보면 5이전에 급격한 기울기 변화가 보이고, 10~15 이후부터는 완만한 형태로 바뀌어 최종적으로 0에 수렴하게 된다.
전체 데이터에 대해 최대한 데이터의 구조를 유지하되, 적은 수의 주성분으로 구성하는 것이 목적이기 때문에
적절한 지점(이 아닐수도 있는)으로 우선 판단되는 10으로 n_components를 설정하여 이후 모델링을 진행해보도록 하자.
pca = PCA(n_components=10)
pca.fit(X_train)
http://bit.ly/3Y34pE0
본 포스팅은 패스트캠퍼스 환급 챌린지 참여를 위해 작성되었습니다.
'Analytics > Study' 카테고리의 다른 글
| 실전#5 미국의 대통령은 어떻게 뽑힐까(7) (0) | 2023.03.21 |
|---|---|
| 실전#5 미국의 대통령은 어떻게 뽑힐까(5) (0) | 2023.03.19 |
| 실전#5 미국의 대통령은 어떻게 뽑힐까(4) (0) | 2023.03.18 |
| 실전#5 미국의 대통령은 어떻게 뽑힐까(3) (0) | 2023.03.17 |
| 실전#5 미국의 대통령은 어떻게 뽑힐까(2) (0) | 2023.03.16 |



